Edwardを試してみた(1) MCMCでベイズ線形回帰
今更ながら確率的プログラミングのライブラリEdwardを試してみました。
http://edwardlib.org/tutorials/
確率的プログラミング
統計的なモデルの(ベイズ)推定が出来るという点でstanやpymcと似ていますが、Edwardはより広く確率的プログラミング(Probabilistic Programming)
という枠組みを実現したライブラリであり、Variable AutoEncoder(VAE)やGenerative adversarial networks(GAN)なども比較的簡単に実装できるようです。
また直接関係ないのですがstanやpymcでは最初にMCMCが実装されあとで自動変分ベイズが実装されたのでインターフェースがMCMCよりですが、EdwardはMCMC,変分ベイズ双方での計算ができ、変分ベイズのほうはより細かい計算の指定が出来るような印象です。
インストール
pip install edward
でインストールできるのですが、tensorflowのバージョンに依存するようです。
それでは厳しいのでanacondaを使いたいですが、現時点ではanacondaでは直接対応していないようで、
conda config --add channels conda-forge conda create -n tfed numpy scipy six tensorflow source activate tfed pip install edward
https://github.com/blei-lab/edward/issues/321
のような簡単なworkaroundでインストールできるそうです。
anaconda cloudでは使えるそうです。
https://anaconda.org/akode/edward
例(ベイズ線形回帰)
最も簡単なmodelの例としてベイズ線形回帰をMCMCで行ったものを試してみました。
SGHMC(Stochastic Gradient Hamilton Monte-Carlo)を使ったものは
にあり、マスタケさんのブログ
【Edward】MCMCの数学的基礎からStochastic Gradient Langevin Dynamicsの実装まで - Gunosyデータ分析ブログ
でも同じ例が紹介されています。
def build_toy_dataset(N, w): D = len(w) x = np.random.normal(0.0, 2.0, size=(N, D)) y = np.dot(x, w) + np.random.normal(0.0, 0.01, size=N) return x, y ed.set_seed(42) N = 40 # number of data points D = 10 # number of features w_true = np.random.randn(D) * 0.5 X_train, y_train = build_toy_dataset(N, w_true) X_test, y_test = build_toy_dataset(N, w_true)
データはnumpyのarrayで渡します。pymcと同じです([2])。
X = tf.placeholder(tf.float32, [N, D]) w = Normal(loc=tf.zeros(D), scale=tf.ones(D)) b = Normal(loc=tf.zeros(1), scale=tf.ones(1)) y = Normal(loc=ed.dot(X, w) + b, scale=tf.ones(N))
モデルとそれを構築する確率分布は([3])で宣言されています。観測データは(ここではX)はtf.placeholderで定義されます。
qw = Empirical(params=tf.Variable(tf.random_normal([T, D])))
qb = Empirical(params=tf.Variable(tf.random_normal([T, 1])))
推定(inference)のオブジェクトを作る段階でMCMC系の方法を使う場合には推定対象の変数をEmpiricalで定義しないといけません([5])。
モデルを構成する変数の確率分布(w,b)と推定対象の分布(qw,qb)は別であるということなのでしょうが観測変数も推定したい変数も(ある意味)2回定義しないといけない書き方がstan,pymcとは少し違って最初戸惑いました*1。
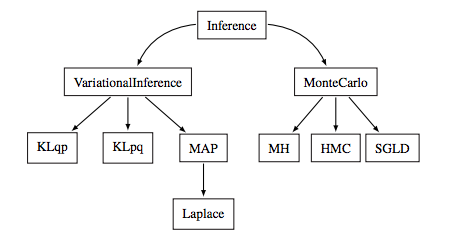
色々なサンプリング、最適化手法の中のひとつひとつとしてMetropolis-Hasting, HMC, SGMCなどは位置づけていて
edward/edward/inferences at master · blei-lab/edward · GitHub
にそれぞれ実装されています。
inference_HMC = ed.HMC({w: qw, b: qb}, data={X: X_train, y: y_train})
inference_HMC.run(step_size=1e-3)
inferenceの定義では引数に観測データに対応する変数dataと同様に推定したい変数(ここではqw,qb)を辞書でw,bとのpairとして渡しています。
inference.run()という書き方は他のMCMCライブラリと似ていますがEdwardではinference.update()を繰り返すような書き方もあるそうで一定の条件を満たしたら止めるような処理も書けるらしいです。
criticism
生成されたデータと正解データを比較してモデルの評価を行うという機械学習でよく行われる方法ですが
y_post=ed.copy(y, {w: qw, b: qb})
でコピーしたy_postとXをテスト用のデータX_test,y_testと組にして
{X: X_test, y_post_HMC: y_test}
ed.evaluateで平均二乗誤差、平均絶対誤差で評価しています([7],[9],[18])。
可視化
stanやpymcではchainやヒストグラムの可視化はmatplotlibなど外部のpythonライブラリを使うのが普通ですが、Edwardではtensorflowの中にあるtensorboardを使っても結果を表示できるそうです*2。
http://edwardlib.org/tutorials/tensorboard